2020年8月20日,清华大学生命科学院陈春来课题组、香港科技大学黄旭辉课题组、北京大学赵新生课题组和中科院福建物质结构研究所张璐研究员合作在《美国科学院院报》(pnas)杂志发表了题为“利用扫描fret-fcs和马尔可夫态模型揭示糖基化酶alkd找寻和识别靶标的机制”(target search and recognition mechanisms of glycosylase alkd revealed by scanning fret-fcs and markov state models)的文章。
dna结合蛋白识别dna上特异性位点是基因复制、dna损伤修复、表达调控和crispr-cas适应性免疫等重要生理生化过程的基础。然而dna结合蛋白如何在包含几百万甚至几十亿个碱基对的基因组中高效并准确的识别其特异性结合位点,仍然是个未解之谜。dna糖基化酶是一类极其重要的dna结合蛋白,作为dna修复酶,它的功能是识别基因组上的dna损伤位点并介导相应的修复过程,在维持基因组稳定性中发挥着重要的功能。捕捉dna糖基化酶在搜寻靶标过程中的动力学信息,对于阐明糖基化酶介导的dna修复过程的分子机制和生理功能都是必不可少的。
人们推测糖基化酶在dna上一维扩散时存在着“高速低准确度”和“低速高准确度”两种靶标搜寻模式,其通过在两种搜寻模式之间切换实现找寻靶标的高效和高准确度。迄今为止,人们并不了解糖基化酶等只存在单一结构域的dna结合蛋白是如何实现在“高速低准确度”和“低速高准确度”搜寻模式之间切换的。究其原因是由于目前测量技术的空间分辨率和时间分辨率有限,研究者仅能直接捕捉到糖基化酶等单结构域蛋白在dna上的高速搜寻模式,而低速搜寻模式以及dna结合蛋白在两种搜寻模式之间的切换过程则没有直接的实验证据。
针对以上技术问题,本文发展了新型高时间分辨率(亚微秒)和高空间分辨率(亚纳米)的扫描荧光共振能量转移相关光谱技术(scanning fret-fcs)。利用这一新技术,他们不但捕捉到糖基化酶alkd在双链dna(dsdna)上的快速一维扩散模式,还首次捕捉到了alkd一维扩散的慢搜寻模式(图1)。从中定量得到快速搜寻过程的扩散系数是8´106 bp2 s-1,这与前人的测量值是相吻合的;而慢速搜寻过程的扩散系数是6´104 bp2 s-1。在此基础上,他们利用大规模的全原子分子动力学模拟结合马尔可夫态模型,进一步揭示了慢搜寻模式的工作机制,发现alkd在沿着dsdna螺旋结构移动一个碱基对的过程中,平动过程和转动过程是依次进行的。alkd相对dsdna的转动较慢,是一维扩散的决速步。利用动力学模型得到的扩散时间尺度与实验测量值很好地吻合,佐证了计算模型的准确性。他们还结合实验测量和动力学模拟,在原子相互作用层面仔细探究了y27位残基对一维扩散过程的影响机制。

图1:融合单分子scanning fret-fcs测量与分子动力学模拟阐释alkd在dsdna上的扩散动力学过程和分子机制。
文章最后提出了如图2所示的模型:在未修饰的正常碱基区域,alkd与dsdna相互作用较弱,此时主要利用快速模式实现高效快速的搜寻;而修饰碱基会导致附近dsdna双螺旋结构的变化,从而诱导alkd-dsdna结合构象的变化和相互作用力的加强,促使alkd从快速转变为慢速搜寻模式,实现对修饰碱基的准确识别。在此模型中,alkd通过改变与dsdna的相互作用构象和结合强弱,实现在快慢两种搜寻模式中的切换和调控,从而获得靶标搜寻中的高效和高准确度。本文发展了紧密融合单分子扫描荧光共振能量转移-相关光谱(scanning fret-fcs)测量与马尔可夫态模型的新型技术平台,突破单一技术手段的局限,为揭示糖基化酶和其它dna结合蛋白搜寻靶标的动态过程和分子机制提供了新的技术平台。
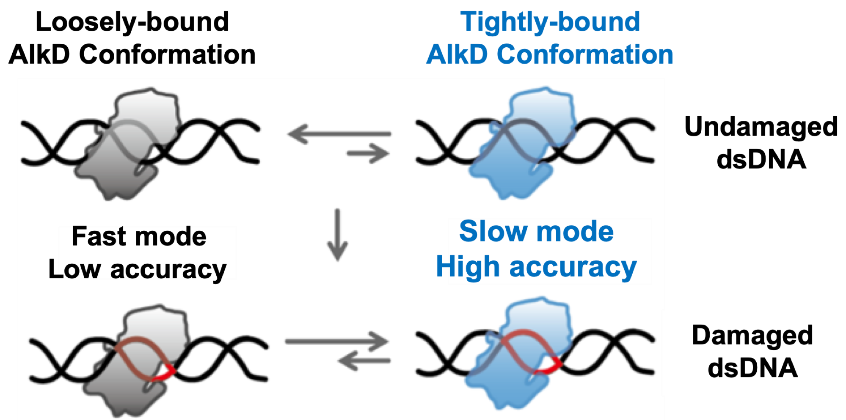
图2:alkd找寻和识别靶标的分子机制。
清华大学生命科学学院陈春来研究员、香港科技大学黄旭辉教授、北京大学赵新生教授和中科院福建物质结构研究所张璐研究员为本文共同通讯作者。清华大学生命学院15级博士生彭思佳、香港科技大学博士生王晓维和中科院福建物质结构研究所张璐研究员为共同第一作者。北京大学博士生何姗珊参与了fret-fcs实验。本工作获得了国家自然科学基金委、北京结构生物学高精尖创新中心、北京生物结构前沿研究中心、清华-北大生命科学联合中心及中国香港研究资助局的经费支持。
原文链接
https://doi.org/10.1073/pnas.2002971117