dna拓扑结构变化可调控r-loop的动态平衡,与基因组稳定性密切相关。dna拓扑异构酶1(top1)作为真核生物高度保守的拓扑异构酶,其特异性抑制剂cpt(camptothecin)可诱导基因组r-loop的积累,并导致基因组不稳定。然而,目前对于基因组拓扑结构变化如何影响r-loop水平进而调控基因组稳态的机制仍然未知。

2023年11月27日,清华大学生命学院孙前文实验室在《自然·通讯》(nature communications) 期刊上发表了题为 “dna聚合酶ε协调基因组拓扑状态和r-loop 的形成以保持拟南芥基因组的完整性” (dna polymerase ε harmonizes topological states and r-loops formation to maintain genome integrity in arabidopsis) 的研究论文,揭示了拟南芥中dna聚合酶ε 参与调控topor-loop动态变化和 dna复制进程,进而维持基因组完整性的分子机制。
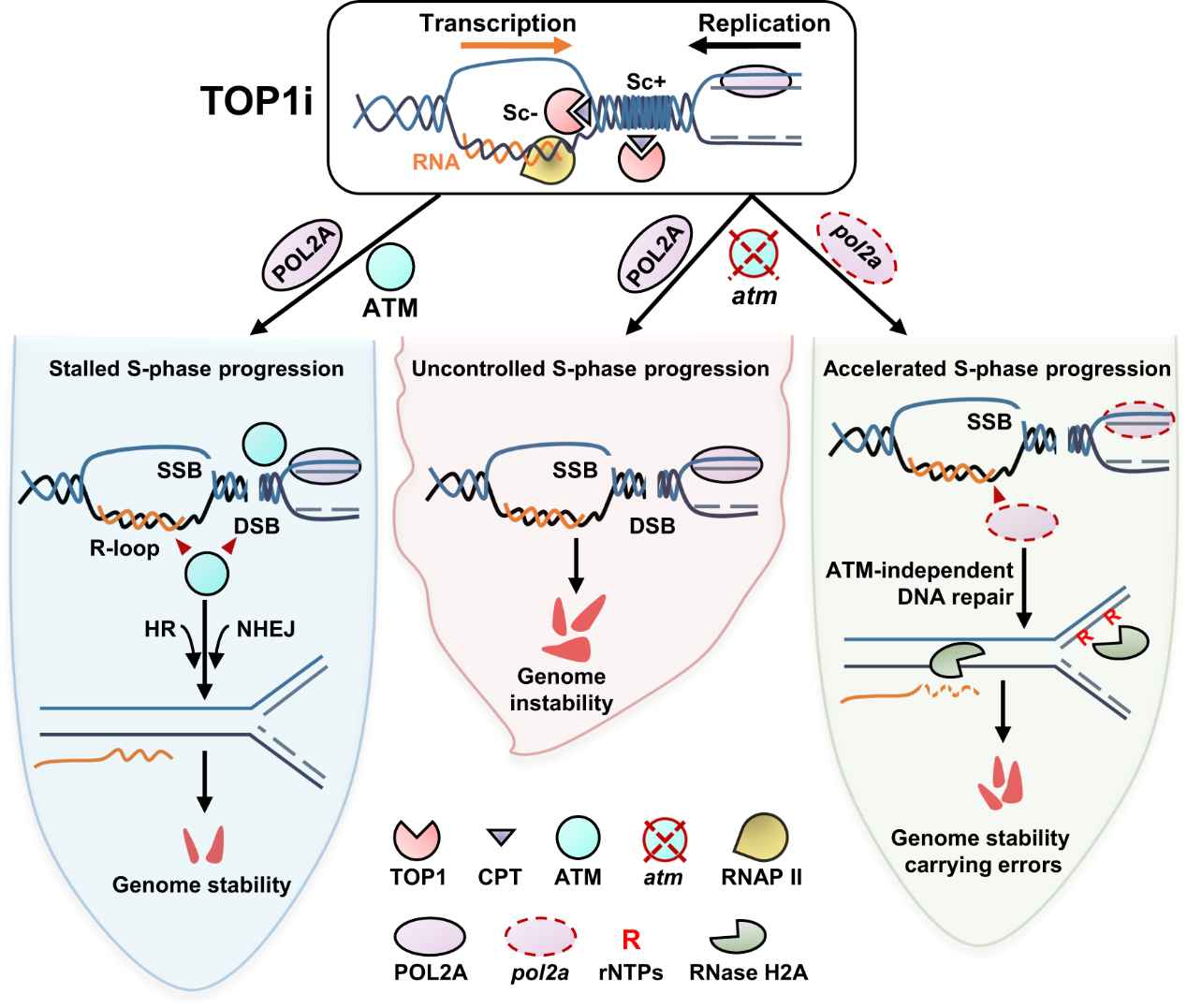
作者首先在拟南芥中应用top1抑制剂(top1i, top1 inhibitor)建立了一个反映r-loop水平与基因组拓扑状态的监测系统,发现cpt处理可显著促进根尖组织中基因组r-loop(拓扑结构发生改变导致的r-loop变化,简称为topor-loop)水平和dna断裂标记γh2ax水平的升高,并最终抑制根生长。利用cpt进行反向遗传筛选发现dna损伤修复激酶atm的突变对cpt尤其敏感,基因组r-loop水平积累尤为显著,根的生长受到严重抑制。在atm突变体基础上利用该系统筛选鉴定出负责先导链合成的dna聚合酶ε催化亚基pol2a的突变,可恢复atm中top1i诱导的topor-loop积累和dna损伤。深入研究发现pol2a可抑制dna复制起始位点附近topor-loop的积累,并降低cpt处理后正在进行dna复制的细胞中的dna损伤水平。该研究表明,dna聚合酶ε可响应基因组拓扑结构变化,协同调控r-loop动态变化和dna复制进程,从而维持基因组的完整性。该发现对深入理解人类癌症化疗过程中atm缺陷导致top1i靶向药物耐药性的机制提供了重要信息,同时为联合使用dna损伤药物和分层治疗提供可能的新策略。
清华大学生命学院2016级已毕业博士李沁是本文第一作者,孙前文副教授为通讯作者。该工作得到了同实验室成员周劲聪博士、李帅博士、张卫峰博士、2017级博士研究生杜盈雪、李宽博士,以及复旦大学王应祥教授的帮助。该研究得到了国家自然科学基金、国家科技部基金、清华-北大生命科学联合中心的资助。
原文链接: