crispr-cas系统是一种广泛应用的基因编辑工具,该系统通过引导rna(grna)引导cas蛋白识别并切割靶标dna。近年来,随着生物信息学和生物化学研究的深入,crispr系统的多样性得到了极大扩展,尤其是在第v型家族中,这类系统依赖于高度保守的ruvc核酸酶结构域来实现靶标切割。作为第v型家族的一个独特亚型,crispr-cas12e(也称为casx)以其较小的分子尺寸和高效的基因编辑潜力而备受关注。已鉴定的dpbcas12e和plmcas12e同源蛋白在蛋白序列和结构方面高度保守。不久之前,清华大学陈春来与刘俊杰(gogo)课题组合作揭示了二者在靶标搜索和切割过程的动态调控机制。然而,crispr-cas12e系统的进化丰富性,以及功能和结构的差异性长期以来缺乏系统性的研究。
2024年12月30日,清华大学生命学院刘俊杰课题组再次联合陈春来课题组以及北京大学现代农业研究院的苏丁丁课题组在《自然-通讯》(nature communications)发表了题为“cas12e同源蛋白进化出多样的结构特征来促进双链dna的解旋”(cas12e orthologs evolve variable structural features to facilitate dsdna cleavage)的研究论文。这项研究系统地鉴定了六种新型cas12e同源蛋白,通过结构生物学和生化实验系统解析了这些蛋白在识别dna靶标、双链dna解旋和切割中的独特机制。
研究团队通过生物信息学分析出六种新的cas12e同源蛋白,其大小在818-920个氨基酸残基(图1)。从预测的结构上分析,这些蛋白的非靶标链结合(ntsb)结构域有明显差别。生化实验表明,cas12e蛋白偏好识别富含t或c的pam序列,并且表现出多样的靶向dna干扰能力。利用单分子fret实验,研究进一步发现,cas12e蛋白的切割效率与盐浓度密切相关。在低盐条件下,这些蛋白表现出更高的r-loop形成能力和切割活性,而高盐条件则会显著抑制其活性(图1)。这一结果揭示了盐浓度对cas12e功能的调控作用,并为其在复杂环境中的应用优化提供了指导。
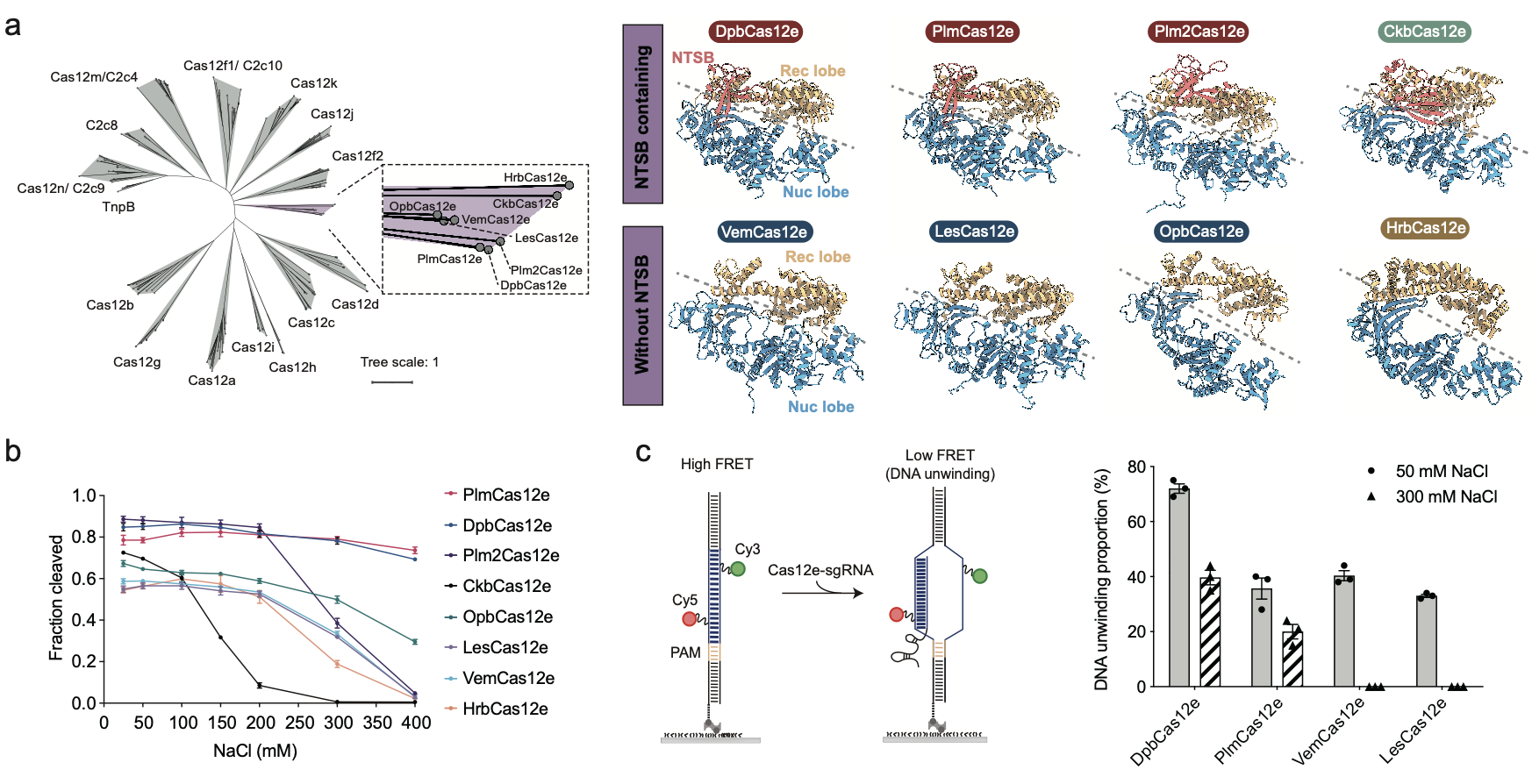
图 1 新型crispr-cas12e系统的结构和生化活性
进一步,课题组通过单颗粒冷冻电镜技术解析了vemcas12e和lescas12e的三元复合体结构。分析发现,在高盐条件下,同源蛋白间不同的r-loop形成能力和cas蛋白中负责解旋dna双链的结构有关(图2)。部分cas12e蛋白(如plmcas12e和dpbcas12e)含有独特ntsb结构域。这一结构域能够在高盐条件下促进dna解旋而增强切割活性。相比之下,缺乏该结构域的cas12e蛋白(如vemcas12e和lescas12e)则依赖富含正电氨基酸残基的短肽环结构(如loop 1和loop 2)进行dna解旋,但其对r-loop形成的促进作用相对较小,因而切割效率相对较低。总之,能够在高盐浓度条件下促进r-loop稳定形成的结构有利于实现更高的dna双链切割活性。与此相符的是,plm2cas12e蛋白的ntsb结构域缺乏一些和非靶标链(nts)、靶标链(ts)相互作用的关键氨基酸残基,因而在高盐浓度的条件下表现出较低的双链切割活性;而opbcas12e蛋白在ruvc核酸酶结构域具有一个能够促进r-loop形成的helix-loop结构,从而在高盐浓度的条件下有相对更高的双链切割活性(图1)。这些结构差异揭示了cas12e家族的功能多样性的结构基础。
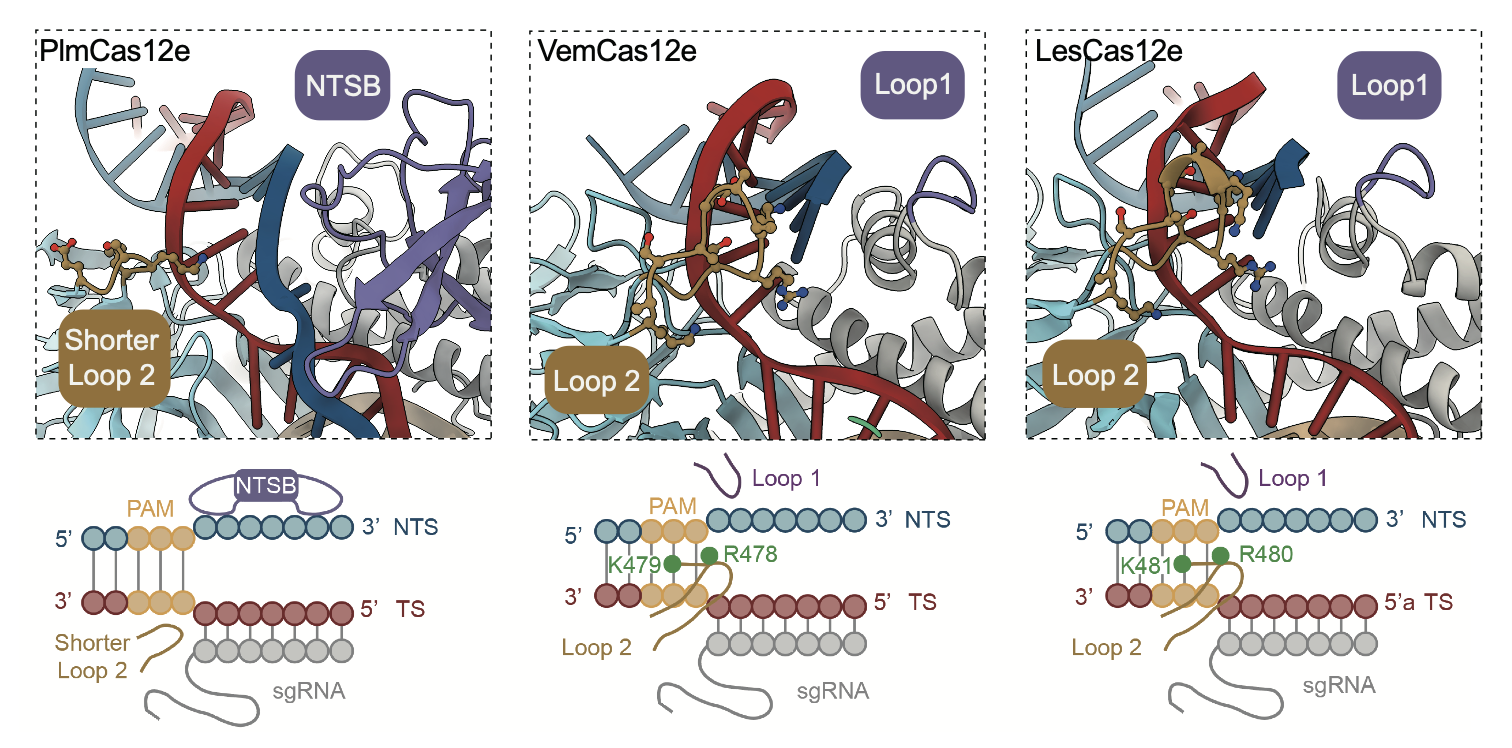
图 2 不同cas12e蛋白参与解旋pam附近双链区域的结构
最后,研究从进化的角度描述了cas12e家族蛋白如何通过获得不同的结构元件,以及相应grna的多样性来适应多样化的环境条件,从而提高其切割活性(图3)。这些发现为未来cas12e系统的优化和基因编辑工具的开发提供了全新思路,也对理解crispr-cas系统的演化机制具有重要意义。
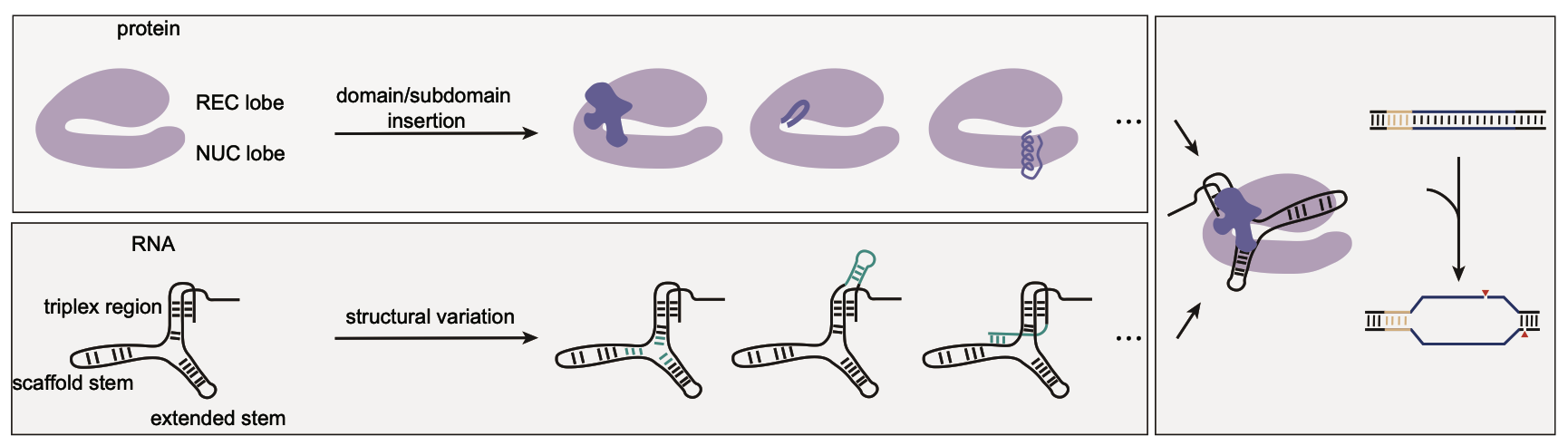
图 3 crispr-cas12e家族的蛋白和引导rna的进化多样性
在本研究中,cas12e同源蛋白展现出了多样的单链dna反式切割活性,其中plmcas12e具有最高效的反式切割活性。当盐浓度降低时,其活性进一步增强。这些结果展现了cas12e家族蛋白在核酸检测中的巨大潜力,并为优化其检测性能提供了理论基础。
清华大学生命学院刘俊杰副教授、陈春来副教授和北京大学现代农业研究院的苏丁丁研究员为本文共同通讯作者。清华大学生命学院2018级博士生李丹苑、博士后张寿悦(现为中科院微生物研究所研究员)、2023级博士生林铄和2018级博士生邢文婧为本文共同第一作者。清华大学生命学院2019级博士生杨韵和北京大学现代农业研究院的2022级硕士生朱凤霞也参与了本研究。该研究得到了国家科技部脑科学项目、国家重点研发计划、中华人民共和国农业农村部、国家自然科学基金等的经费支持。
相关文章链接: